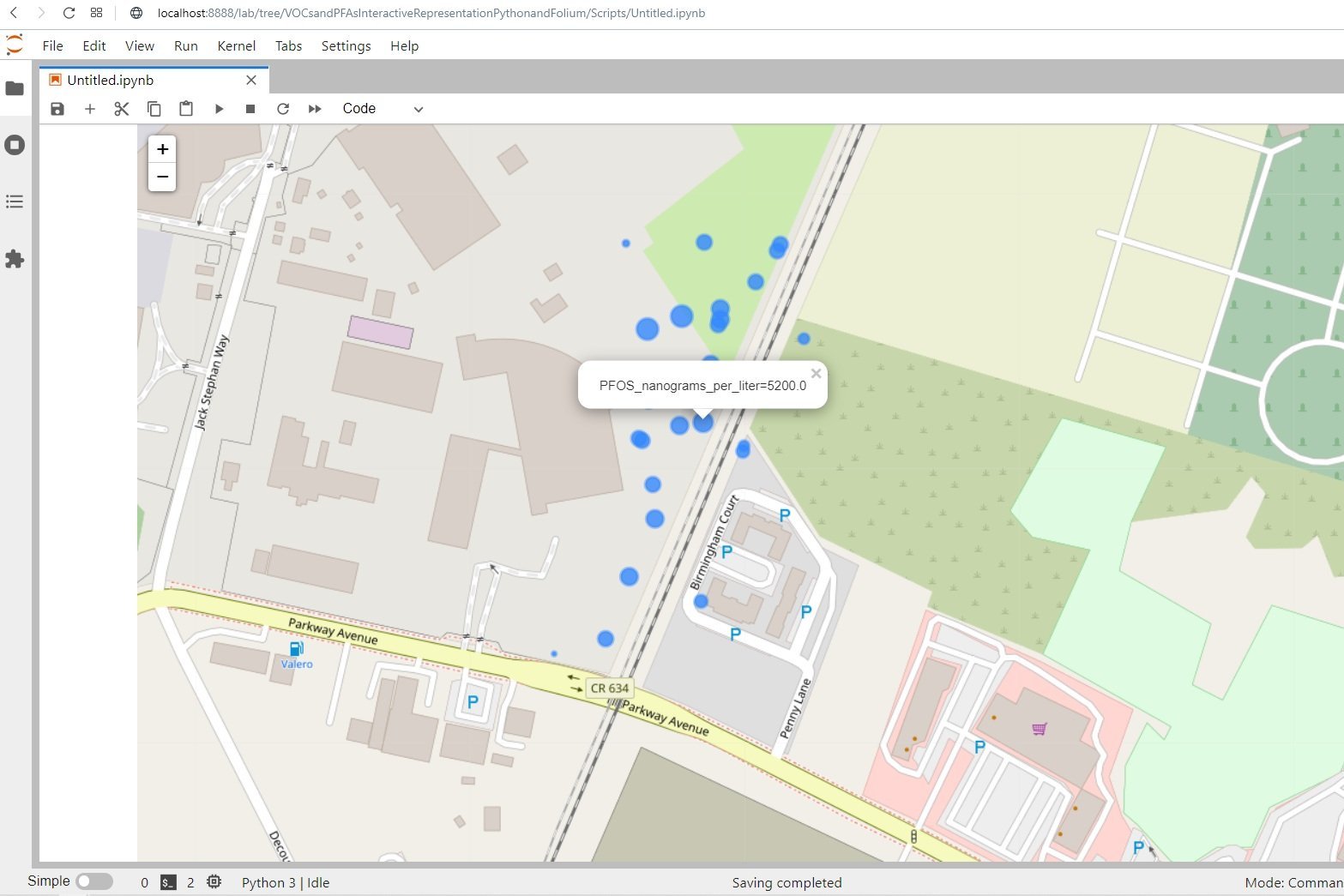
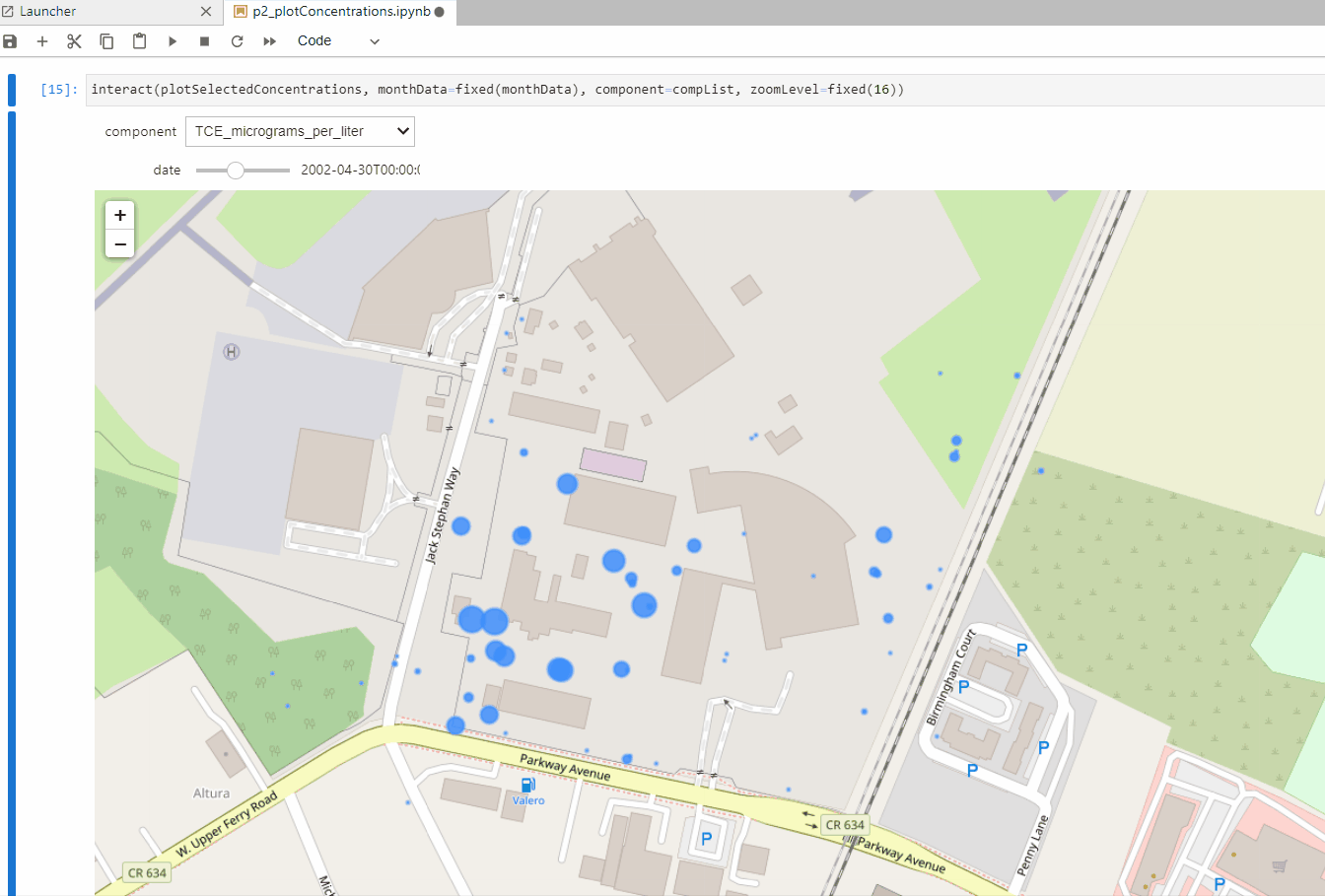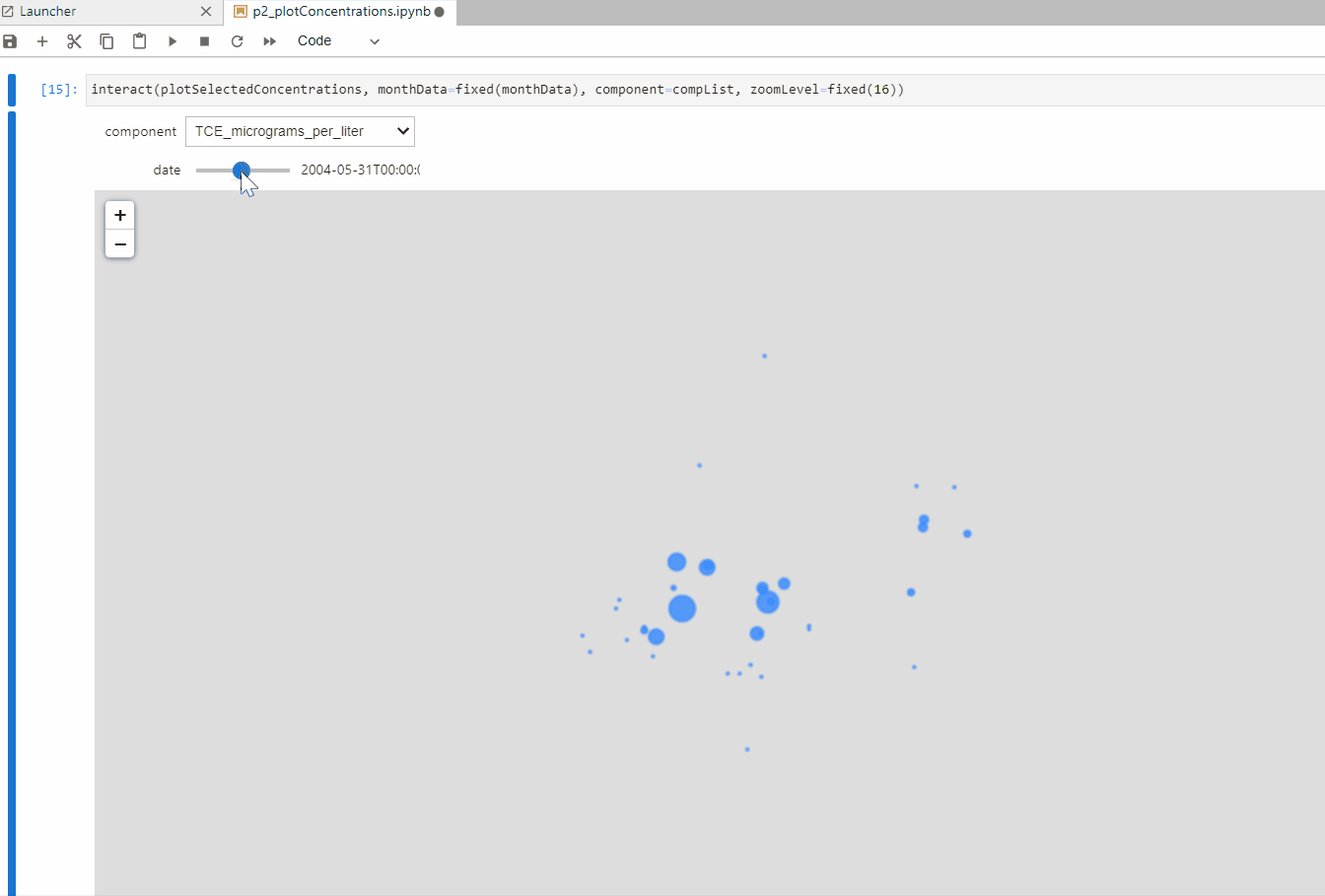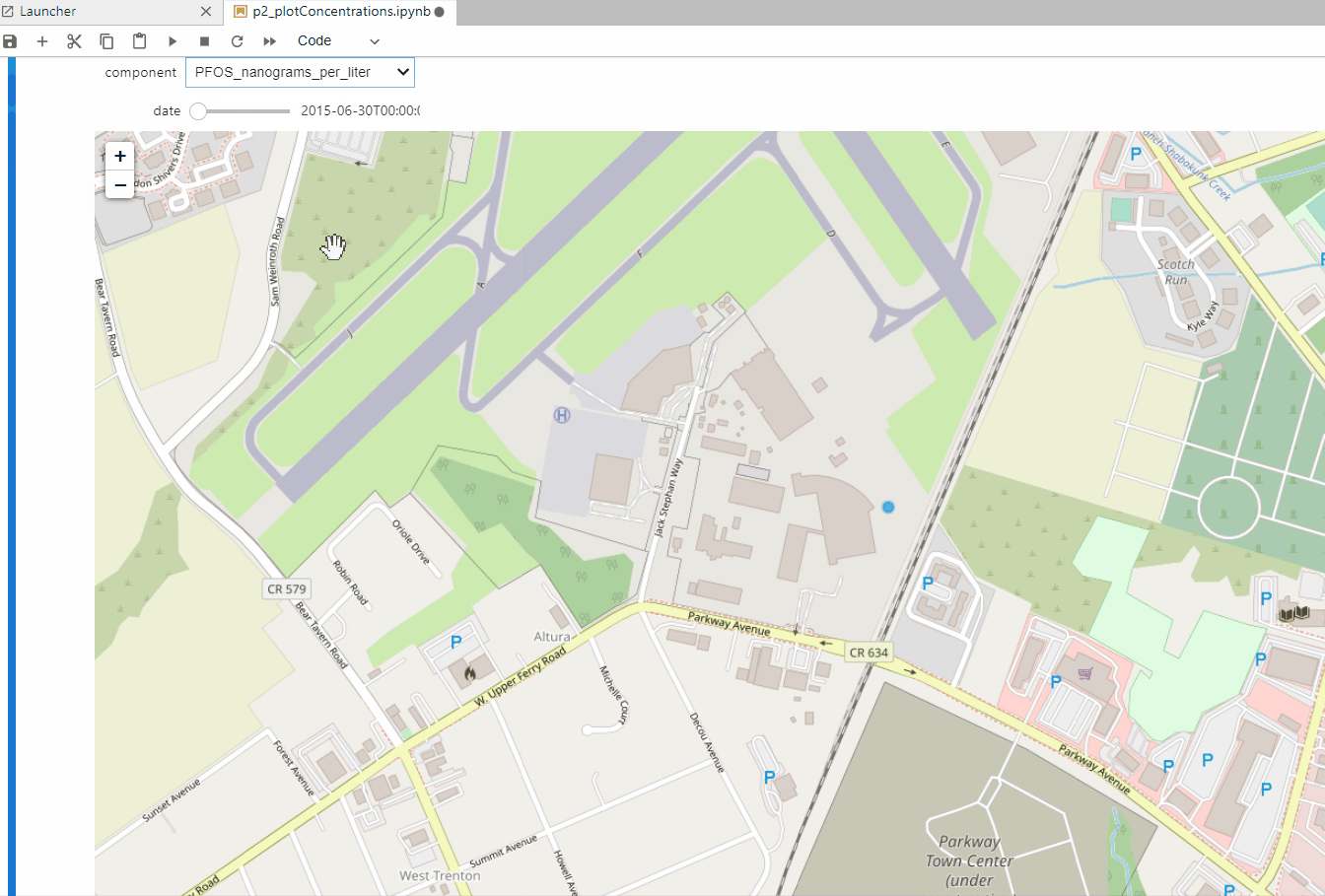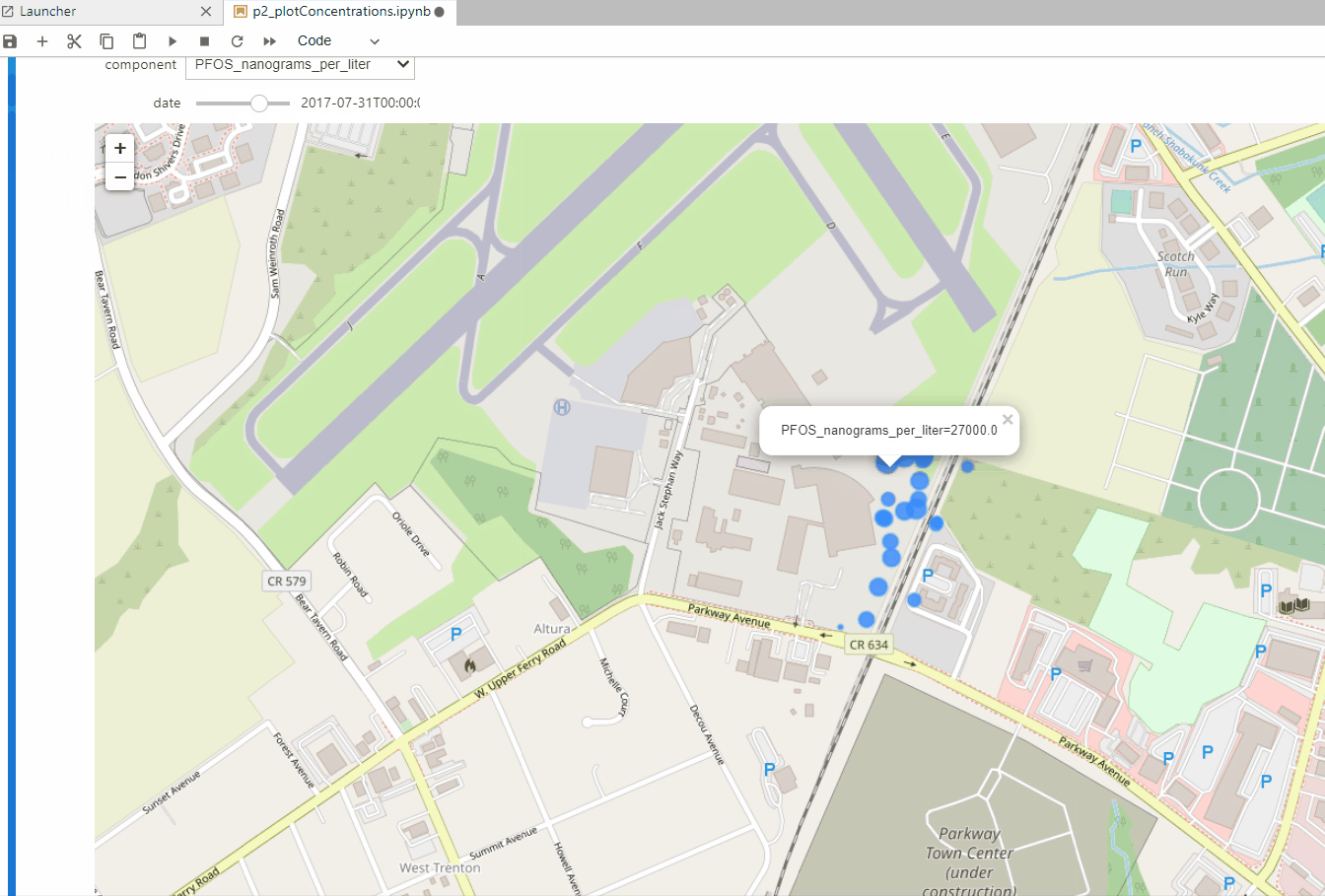
El análisis de la química del agua subterránea es una tarea difícil para el conjunto limitado de puntos de observación, las muestras limitadas y los limitados componentes analizados. Para evaluar la extensión real de una pluma de contaminación o la eficiencia de las técnicas de remediación necesitamos métodos nuevos e innovadores que permitar representar y analizar los datos de la química del agua con software libre. Hemos realizado un caso aplicado de representación interactiva de VOC y PFA en un Jupyter notebook con Python, Folium e Ipywidgets. El conjunto de datos tiene más de 3300 muestras de 127 puntos durante un período de 30 años y corresponde a un sitio contaminado de un antiguo aeropuerto.
Instale Ipywidgets en Jupyterlab siguiendo las instrucciones de este enlace:
ipywidgets.readthedocs.io/en/latest/user_install.html
Video
Animación
Código
import numpy as np
import pandas as pd
import folium
from ipywidgets import interact, fixed, SelectionSliderobsLoc = pd.read_csv('../Csv/obsLoc_v2.csv', index_col=0)
obsLoc.head()obsData = pd.read_csv('../Txt/NAWC_GWquality_Filter.txt',delimiter='\t',
parse_dates=[3], na_values=['NA','NS','NR'])
obsData.dtypesmIndex = pd.MultiIndex.from_frame(obsData[['NWIS_SiteNumber','SampleDate']],
names=['Number','Date'])
obsData.index = mIndex
obsData.head()#get values for map location
lonMean = obsLoc.lon.mean()
latMean = obsLoc.lat.mean()
print(latMean,lonMean)#show all monitoring points
m = folium.Map(location=[latMean,lonMean],zoom_start=15)
for index,row in obsData.iterrows():
try:
lat, lon = obsLoc.loc[index[0]].values
folium.CircleMarker(location=[lat, lon], radius=2).add_to(m)
except KeyError:
pass
m# sample of group samples by month
monthData = obsData.groupby(by=['NWIS_SiteNumber',pd.Grouper(freq='M', level=1)]).mean()
monthData.iloc[[0]]#list of sampling dates
uniqueDates=pd.unique(monthData.index.get_level_values(1))
uniqueDates = np.sort(uniqueDates)
uniqueDates.shape#list of components
compList = [a for a in monthData.keys().to_list() if 'per_liter' in a]
compList# filter dataframe by component values
monthData[monthData['PFUnA_nanograms_per_liter']>0]#plot function for given time
def plotConcentrations(monthData,component,date,zoomLevel):
#initalize map
m = folium.Map(location=[latMean,lonMean],zoom_start=zoomLevel)
#filter by date:
tempDf = monthData[monthData.index.get_level_values(1) == date]
tempDf = tempDf[[component]]
#maxCon = monthData[component].max()
for index,row in tempDf.iterrows():
try:
lat, lon = obsLoc.loc[index[0]].values
folium.CircleMarker(location=[lat, lon],
radius=np.log(row[component]+0.001),
popup=component +'='+str(row[component]),
opacity=0.4,
fill=True,
fill_opacity=0.8).add_to(m)
except KeyError:
pass
return m#plot concentration for a given date
plotConcentrations(monthData,'VC_micrograms_per_liter',uniqueDates[30],zoomLevel=15)#plot function for given time
def plotSelectedConcentrations(monthData,component,zoomLevel):
tempDf = monthData[monthData[component]>0]
uniqueDates=pd.unique(tempDf.index.get_level_values(1))
uniqueDates = np.sort(uniqueDates)
interact(plotConcentrations, monthData=fixed(monthData), component=fixed(component),
date=SelectionSlider(options=list(uniqueDates)),
zoomLevel=fixed(zoomLevel));# plot one component
plotSelectedConcentrations(monthData,'PFOS_nanograms_per_liter',16)interact(plotSelectedConcentrations, monthData=fixed(monthData), component=compList, zoomLevel=fixed(16))Datos de Ingreso
Puede descargar los datos de entrada desde este enlace.